
réaction en chaîne de la polymérase
Saviez-vous ...
SOS croit que l'éducation donne une meilleure chance dans la vie des enfants dans le monde en développement aussi. Mères SOS chaque regard après une une famille d'enfants parrainés .
La réaction en chaîne par polymerase (PCR) est une technique largement utilisée dans biologie moléculaire. Il tire son nom de l'un de ses éléments clés, un ADN polymérase utilisée pour amplifier un fragment de l'ADN par in vitro enzymatique réplication. Comme progresse PCR, l'ADN ainsi produit est lui-même utilisé comme matrice pour la réplication. Cela déclenche une réaction en chaîne dans laquelle la matrice d'ADN est exponentielle amplifié. Avec la PCR, il est possible d'amplifier une seule ou quelques copies d'un fragment d'ADN dans plusieurs ordres de grandeur, des millions de génération ou de plusieurs copies du morceau d'ADN. La PCR peut être largement modifié pour exécuter un large éventail de manipulations génétiques.
La quasi-totalité des applications de PCR utilisent une ADN polymerase thermostable, telle que Taq polymérase, une enzyme isolée à l'origine à partir de la bactérie Thermus aquaticus. Cet ADN polymérase enzymatique assemble un nouveau brin d'ADN à partir de blocs de construction ADN, le nucleotides, en utilisant l'ADN simple brin comme matrice et oligonucleotides d'ADN (également appelés des amorces d'ADN) nécessaires pour l'initiation de la synthèse d'ADN. La grande majorité des méthodes PCR utilisation cyclage thermique, ce est à dire, alternativement chauffer et refroidir l'échantillon de PCR à une série définie d'étapes de température. Ces étapes thermiques de cyclisme est nécessaire de séparer physiquement les brins (à haute température) dans une double hélice d'ADN ( ADN fusion) utilisé comme modèle lors de la synthèse d'ADN (à des températures inférieures) par l'ADN polymérase pour amplifier sélectivement l'ADN cible. La sélectivité de PCR résultant de l'utilisation de des amorces qui sont complémentaire de la région d'ADN ciblée pour l'amplification dans des conditions thermiques spécifiques de cyclisme.
Développé en 1983 par Kary Mullis, PCR est maintenant une technique courante et souvent indispensable utilisé dans les laboratoires de recherche médicale et biologique pour une variété d'applications. Il se agit notamment clonage de l'ADN pour basé sur le séquençage d'ADN, phylogénie, ou analyse fonctionnelle des gènes; le diagnostic de maladies héréditaires; l'identification de empreintes génétiques (utilisés dans sciences médico-légales et les tests de paternité); et la détection et le diagnostic de maladies infectieuses . En 1993 Mullis a remporté le prix Nobel de chimie pour ses travaux sur la PCR.
Principes et la procédure PCR


La PCR est utilisée pour amplifier des régions spécifiques d'un brin d'ADN (l'ADN cible). Il peut se agir d'un seul gène, une partie d'un gène ou une séquence non codante. La plupart des méthodes de PCR amplifient généralement des fragments d'ADN allant jusqu'à 10 paires de bases (kb kilo), bien que certaines techniques permettent une amplification de fragments jusqu'à 40 kb.
Une PCR base mis en place nécessite plusieurs composants et réactifs. Ces composants incluent:
- matrice d'ADN qui contient la région d'ADN (cible) à amplifier.
- Deux amorces, qui sont complémentaires des régions d'ADN à la 5 '(cinq prime) ou 3 '(de trois prime) extrémités de la région d'ADN.
- Un ADN polymerase telle que La polymerase Taq ou une autre polymerase d'ADN avec une température optimale autour de 70 ° C.
- Désoxynucléosides triphosphates (dNTP; aussi très souvent et à tort appelés désoxynucléotide triphosphates), les blocs de construction à partir de laquelle les ADN polymérases synthétise un nouveau brin d'ADN.
- Solution tampon, fournissant un environnement chimique approprié pour une activité optimale et la stabilité de l'ADN polymérase.
- Bivalents cations , magnésium ou de manganèse ions; généralement on utilise Mg 2+, Mn 2+, mais peut être utilisé pour la mutagenèse médiée par PCR de l'ADN, comme la concentration plus élevée en Mn 2+ augmente le taux d'erreur au cours de la synthèse d'ADN
- Cation monovalent potassium ions.
La PCR est généralement effectuée dans un volume réactionnel de 10 à 200 ul dans de petits tubes de réaction (0,2 à 0,5 ml volumes) dans un thermocycleur. Le cycleur thermique chauffe et refroidit les tubes de réaction pour atteindre les températures nécessaires à chaque étape de la réaction (voir ci-dessous). De nombreux thermocycleurs modernes font usage de la Effet Peltier qui permet le chauffage et le refroidissement du bloc de maintien des tubes de PCR en inversant simplement le courant électrique. Tubes de réaction à parois minces permettent conductivité thermique favorable pour permettre l'équilibrage thermique rapide. La plupart des thermocycleurs ont couvercles chauffants pour éviter la condensation au sommet du tube de réaction. Thermocycleurs âgées dépourvues de couvercle chauffant nécessitent une couche d'huile sur le dessus du mélange de réaction ou une boule de cire à l'intérieur du tube.
Procédure
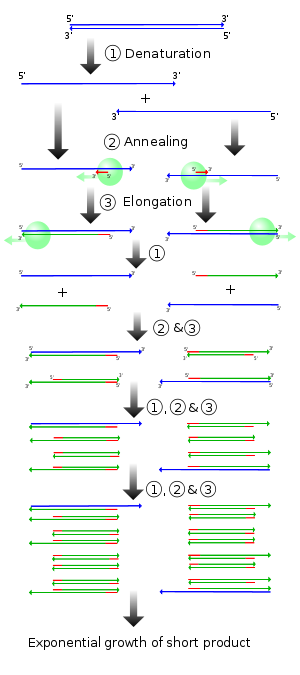
La PCR est généralement constitué d'une série de 20 à 40 les variations de température répétées appelées cycles; chaque cycle se compose généralement de deux à trois niveaux de température distincts. Le plus souvent, la PCR est effectuée avec des cycles ayant trois paliers de température (fig. 2). Le vélo est souvent précédée par une étape de température unique (appelé de maintien) à une température élevée (> 90 ° C), et suivi par une attente à la fin de l'extension du produit final ou une brève stockage. Les températures utilisées et de la durée pendant laquelle ils sont appliqués dans chaque cycle dépendent d'une variété de paramètres. Ceux-ci comprennent l'enzyme utilisée pour la synthèse de l'ADN, la concentration des ions divalents et des dNTP à la réaction, et la température de fusion ( Tm) des amorces.
- l'étape d'initialisation: Cette étape consiste à chauffer la réaction à une température de 94-96 ° C (ou 98 ° C si polymerases thermostables sont extrêmement employées), qui est maintenu à 1-9 minutes. Il est seulement nécessaire pour les ADN polymérases qui nécessitent l'activation de la chaleur par PCR de démarrage à chaud .
- étape de dénaturation: Cette étape est le premier événement cycliste régulier et consiste à chauffer la réaction à 94-98 ° C pendant 20-30 secondes. Il provoque la fusion de la matrice d'ADN et des amorces de rupture des liaisons hydrogène entre les bases complémentaires des brins d'ADN, ce qui donne des simples brins d'ADN.
- l'étape de recuit: La température de réaction est abaissée à 50-65 ° C pendant 20 à 40 secondes permettant hybridation des amorces à la matrice d'ADN simple brin. Typiquement, la température de recuit est d'environ 3 à 5 degrés Celsius en dessous de la Tm des amorces utilisées. Liaisons hydrogène ADN-ADN stable ne se forment lorsque la séquence de l'amorce correspond de très près la séquence modèle. La polymerase se lie à l'hybride amorce-matrice et commence la synthèse d'ADN.
- Extension / allongement étape: La température à cette étape dépend de la polymérase d'ADN utilisée; Taq polymerase a son optimum température de l'activité à 75 à 80 ° C, et généralement à une température de 72 ° C est utilisé avec cette enzyme. A ce stade de l'ADN polymérase synthétise un nouveau brin d'ADN complémentaire au brin matrice d'ADN par addition de dNTP qui sont complémentaires de la matrice en 5 'à 3', 5'-condensation de la groupe phosphate des dNTP avec la 3' un groupe hydroxyle à l'extrémité de la naissante (extension) de brin d'ADN. Le temps d'extension dépend à la fois l'ADN polymérase utilisée et de la longueur du fragment d'ADN à amplifier. En règle empirique, à sa température optimale, l'ADN polymérase va polymériser un millier de bases par minute. Dans des conditions optimales, à savoir, se il n'y a pas de limitations dues à la limitation des substrats ou des réactifs, à chaque étape d'extension, la quantité d'ADN cible est doublée, ce qui conduit à exponentielle (géométrique) l'amplification du fragment d'ADN spécifique.
- Allongement final: Cette étape unique est parfois effectuée à une température de 70-74 ° C pendant 5 à 15 minutes après le dernier cycle de PCR pour se assurer que tout l'ADN simple brin restant est complètement étendu.
- Attente final: Cette étape à 4-15 ° C pour une durée indéterminée peut être utilisé pour le stockage à court terme de la réaction.
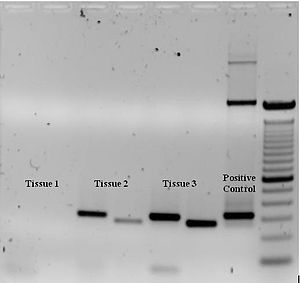
Pour vérifier si le PCR a généré le fragment d'ADN prévu (parfois dénommé amplimer ou amplicon), une électrophorèse sur gel d'agarose est utilisé pour la séparation par taille des produits de PCR. La taille (s) de produits de PCR est déterminée par comparaison avec une échelle d'ADN (un marqueur de poids moléculaire), qui contient des fragments d'ADN de taille connue, exécuter sur le gel à côté des produits de PCR (voir Fig. 3).
Stades PCR
Le procédé de PCR peut être divisé en trois étapes:
Amplification exponentielle: A chaque cycle, la quantité de produit est doublée (en supposant 100% d'efficacité de la réaction). La réaction est très spécifique et précis.
Mise à niveau hors de la scène: la réaction ralentit aussi l'ADN polymérase perd son activité et que la consommation de réactifs tels que dNTP et des amorces les amène à devenir limitante.
Plateau: Pas plus produit se accumule en raison de l'épuisement des réactifs et des enzymes.
Optimisation PCR
Dans la pratique, la PCR peut échouer pour diverses raisons, en partie en raison de sa sensibilité à la contamination de produits provoquant l'amplification d'ADN parasites. De ce fait, un certain nombre de techniques et procédures ont été développées pour l'optimisation des conditions de PCR. La contamination par de l'ADN étranger est adressée aux protocoles et procédures qui séparent mélanges pré-PCR à partir d'ADN contaminants potentiels laboratoire. Cela implique généralement la séparation spatiale des zones PCR-configuration des zones pour l'analyse ou la purification des produits de PCR, et nettoyer à fond la surface de travail entre les configurations de réaction. Primer techniques de conception sont importantes pour améliorer le rendement en produit de PCR et en évitant la formation de produits parasites, et l'utilisation de composants tampon de remplacement ou des enzymes polymerases peuvent contribuer à l'amplification des régions longues ou autrement problématiques de l'ADN.
Application de la PCR
Isolement de l'ADN génomique
PCR permet l'isolement de fragments d'ADN de l'ADN génomique par amplification sélective d'une région spécifique de l'ADN. Cette utilisation de la PCR augmente de nombreuses méthodes, telles que la génération sondes d'hybridation pour Sud ou l'hybridation du Nord et clonage de l'ADN, ce qui nécessite de plus grandes quantités d'ADN, ce qui représente une région spécifique de l'ADN. PCR fournit ces techniques avec des quantités élevées d'ADN pur, permettant l'analyse d'échantillons d'ADN, même de très petites quantités de matière de départ.
D'autres applications de PCR comprennent Le séquençage d'ADN pour déterminer des séquences de PCR amplifié inconnus dans lequel l'une des amorces d'amplification peuvent être utilisées dans le séquençage de Sanger, l'isolement d'une séquence d'ADN à accélérer technologies d'ADN recombinant comportant l'insertion d'une séquence d'ADN dans un plasmide ou le matériel génétique d'un autre organisme. Les colonies bactériennes ( E. coli) peut être projeté rapidement par PCR pour l'ADN correcte constructions de vecteur. PCR peut également être utilisée pour empreintes génétiques; une technique de médecine légale utilisé pour identifier une personne ou organisme en comparant des ADN par différentes méthodes expérimentales basées sur la PCR.
Méthodes «empreintes digitales» Certains PCR ont haute pouvoir discriminant et peuvent être utilisés pour identifier les relations génétiques entre les individus, comme parent-enfant ou entre frères et sœurs, et sont utilisés dans les tests de paternité (Fig. 4). Cette technique peut également être utilisée pour déterminer les relations évolutives entre les organismes.

L'amplification et la quantification de l'ADN
Parce PCR amplifie les régions de l'ADN qu'il cible, la PCR peut être utilisée pour analyser de très petites quantités d'échantillons. Ce est souvent critique pour analyse médico-légale, alors que seulement une trace d'ADN est disponible à titre de preuve. PCR peut également être utilisé dans l'analyse de l'ADN ancien qui est des milliers d'années. Ces techniques basées sur la PCR ont été utilisées avec succès sur des animaux, tels que quarante mille ans de mammouth , et aussi sur l'ADN humain, dans des applications allant de l'analyse des égyptiens momies à l'identification d'une Russie Tsar.
Méthodes de PCR quantitatives permettent l'estimation de la quantité d'une séquence donnée présente dans un échantillon - une technique souvent appliquée pour déterminer quantitativement les niveaux de l'expression du gène. PCR en temps réel est mis en place un outil pour la quantification de l'ADN qui mesure l'accumulation de produit d'ADN après chaque cycle d'amplification PCR.
- Voir également L'utilisation de l'ADN dans l'entomologie médico-légale
PCR dans le diagnostic de maladies
PCR permet un diagnostic précoce des maladies malignes comme la leucémie et les lymphomes, qui est actuellement le plus développé dans la recherche sur le cancer et est déjà utilisé régulièrement. Des dosages de PCR peuvent être réalisées directement sur des échantillons d'ADN génomique à détecter des cellules malignes spécifiques à translocation à une sensibilité qui est au moins 10 000 fois plus élevée que les autres méthodes.
PCR permet également l'identification des micro-organismes non cultivables ou à croissance lente tels que les mycobactéries, des bactéries anaérobies ou des virus provenant de dosages de cultures tissulaires et des modèles animaux. La base de PCR pour des applications de diagnostic en microbiologie est la détection d'agents infectieux et de la discrimination de non-pathogène de souches pathogènes en vertu de gènes spécifiques.
Viral DNA peut également être détecté par PCR. Les amorces utilisées doivent être spécifiques pour des séquences cibles dans l'ADN d'un virus, et la PCR peuvent être utilisées pour des analyses de diagnostic ou de séquençage de l'ADN du génome viral. La grande sensibilité de la PCR permet la détection des virus peu après l'infection, et même avant le début de la maladie. Cette détection précoce peut donner aux médecins une avance significative dans le traitement. La quantité de virus ("charge virale") chez un patient peut également être quantifiée par des techniques d'ADN quantification basées sur la PCR (voir ci-dessous).
Des variantes de la technique PCR de base
- PCR allèle-spécifique: Cette technique de diagnostic ou de clonage est utilisé pour identifier ou exploiter polymorphismes d'un seul nucléotide (SNP) (différences de base unique dans l'ADN). Il nécessite une connaissance préalable de la séquence d'ADN, y compris les différences entre allèles, et utilise des amorces dont extrémités 3 'englober la SNP. L'amplification par PCR dans des conditions stringentes est beaucoup moins efficace en présence d'un mésappariement entre matrice et l'amorce, de sorte amplification réussie d'un signal d'amorces spécifique SNP-SNP de la présence d'une séquence spécifique. Voir Génotypage SNP pour plus d'informations.
- Assemblée PCR ou Polymerase Assemblée Cyclisme (PCA): Assemblage PCR est la synthèse artificielle de longues séquences d'ADN en effectuant une PCR sur un bassin de longs oligonucleotides avec de courts segments qui se chevauchent. Les oligonucléotides alternent entre directions sens et antisens, et les segments se chevauchent déterminent l'ordre des fragments PCR produisant ainsi sélectivement le produit final d'ADN longtemps.
- La PCR asymétrique: PCR asymétrique est utilisée pour amplifier préférentiellement un brin de l'original ADN plus que l'autre. Il trouve une utilisation dans certains types de séquençage et l'hybridation de sondage où avoir un seul des deux brins complémentaires est nécessaire. PCR est effectuée comme d'habitude, mais avec un grand excès des amorces pour le volet choisi. En raison de la lente ( arithmétique ) amplification ultérieure dans la réaction après l'amorce limitante est épuisée, les cycles supplémentaires de PCR sont nécessaires. Une modification récente sur ce processus, connu sous le nom L inear- Un fter- T il- E xponential-PCR (PCR LATE), utilise une amorce limitante avec une température de fusion supérieure ( Température de fusion | Tm) de l'amorce en excès pour maintenir l'efficacité de la réaction lorsque la concentration en amorce limitante diminue mi-réaction.
- Hélicase dépendante de l'amplification: Cette technique est similaire à une PCR classique, mais utilise plutôt une température constante de vélo à travers des cycles de dénaturation et d'annelage / extension. ADN hélicase, une enzyme qui se déroule à l'ADN, est utilisée à la place de la dénaturation thermique.
- Hot-Start PCR: Ce est une technique qui réduit l'amplification non spécifique au cours de la configuration initiale étapes de la PCR. La technique peut être réalisée manuellement en chauffant les composants de la réaction à la température de fusion (par exemple, 95 ° C) avant d'ajouter la polymerase. Systèmes enzymatiques spécialisés ont été mis au point qui inhibent l'activité de la polymérase à température ambiante, soit par la liaison d'un anticorps ou par la présence d'inhibiteurs liés par covalence qui ne se dissocient après une étape d'activation à haute température. Démarrage à chaud / froid finition PCR est réalisée avec de nouvelles polymérases hybrides qui sont inactifs à la température ambiante et sont instantanément activées à la température d'élongation.
- Intersequence spécifique (ISSR) PCR: une méthode PCR pour l'ADN des empreintes digitales qui amplifie les régions entre certaines répétitions de séquences simples pour produire une empreinte digitale unique de longueur des fragments amplifiés.
- PCR inverse: une méthode utilisée pour permettre PCR lorsque seulement une séquence interne est connue. Ceci est particulièrement utile pour identifier des séquences flanquantes aux divers inserts génomiques. Il se agit d'une série de digestions d'ADN et auto-ligature, résultant en des séquences connues aux deux extrémités de la séquence inconnue.
- La ligature de PCR médiée par: Cette méthode utilise de petits lieurs d'ADN lié à l'ADN d'intérêt et de multiples amorces recuit à des lieurs d'ADN; il a été utilisé pour le séquençage d'ADN, le génome marche, et ADN empreinte.
- Spécifique de la méthylation PCR (MSP): La méthode MSP a été développé par Stephen Baylin et Jim Herman à l'École de médecine Johns Hopkins, et est utilisé pour détecter la méthylation des îlots CpG dans l'ADN génomique. ADN est d'abord traité avec du bisulfite de sodium, qui convertit des bases cytosine non méthylées en uracile, qui est reconnu par des amorces de PCR comme thymine. Deux PCR sont ensuite effectués sur l'ADN modifié, en utilisant des ensembles d'amorces identiques sauf à des îlots CpG dans les séquences d'amorces. A ces points, une série d'amorces reconnaît ADN avec cytosines pour amplifier l'ADN méthylé, et un ensemble reconnaît l'ADN avec de l'uracile ou la thymine pour amplifier l'ADN non méthylé. MSP utilisant qPCR peut également être effectuée pour obtenir des informations quantitatives plutôt que qualitatives sur la méthylation.
- Miniprimer PCR: PCR Miniprimer utilise une nouvelle polymerase thermostable (S-ROC) qui peut se étendre à partir de courtes amorces ("smalligos») aussi court que 9 ou 10 nucleotides, au lieu des 20 nucleotides environ requises par Taq. Cette méthode permet de PCR ciblant les régions de liaison d'amorce plus petit, et est particulièrement utile pour amplifier inconnue, mais conservée, des séquences d'ADN, tels que 16S ou 18S (eucaryotes) gène de l'ARNr. ARNr 16S miniprimer PCR a été utilisée pour caractériser une communauté de tapis microbiens croissante dans un environnement extrême, un étang hypersalin à Puerto Rico. Dans cette étude, profondément séquences divergentes ont été découverts à haute fréquence et des représentants que dé fi ni deux nouveaux taxons au niveau de la division, ce qui suggère que la PCR miniprimer peut révéler de nouvelles dimensions de la diversité microbienne. En élargissant "l'espace de séquence» qui peut être interrogé par des amorces de PCR, cette technique peut permettre de nouvelles stratégies de PCR qui ne sont pas possibles dans les limites de conception d'amorce imposées par Taq et d'autres enzymes couramment utilisées.
- Multiplex ligature dépendant Probe Amplification (MLPA): permet de multiples cibles à amplifier avec seulement une seule paire d'amorces, évitant ainsi les limites de résolution de PCR multiplex (voir ci-dessous).
- Multiplex PCR: L'utilisation de multiples jeux d'amorces uniques au sein d'un mélange unique de PCR pour produire amplicons de tailles variables spécifiques pour différentes séquences d'ADN. En ciblant de multiples gènes à la fois, des informations supplémentaires peuvent être obtenues à partir d'un simple test qui nécessiteraient autrement plusieurs fois les réactifs et plus de temps à effectuer. Les températures d'hybridation pour chacun des ensembles d'amorces doivent être optimisées pour fonctionner correctement dans une seule réaction, et tailles des amplicons, ce est à dire, la longueur de la paire de bases, doivent être suffisamment différents pour former des bandes distinctes lorsque visualisé par électrophorèse sur gel.
- PCR nichée: augmente la spécificité de l'amplification d'ADN, en réduisant fond dû à l'amplification non spécifique de l'ADN. Deux jeux d'amorces sont utilisées dans deux PCR successives. Dans la première réaction, une paire d'amorces est utilisée pour générer des produits d'ADN qui, en plus de la cible visée, peut encore consister en des fragments d'ADN amplifiés de manière non spécifique. Le produit (s) sont ensuite utilisés dans une seconde PCR avec un jeu d'amorces dont les sites de liaison sont complètement ou partiellement différente de et situé en 3 'de chacune des amorces utilisées dans la première réaction. PCR nichée est souvent plus réussi à amplifier spécifiquement longs fragments d'ADN que la PCR conventionnelle, mais il nécessite une connaissance plus détaillée des séquences cibles.
- PCR de chevauchement-extension: est un technique de génie génétique permettant la construction d'une séquence d'ADN avec une altération inséré au-delà de la limite de la plus grande longueur de l'amorce pratique.
- PCR quantitative (Q-PCR): est utilisé pour mesurer la quantité d'un produit PCR (de préférence en temps réel). Ce est la méthode de choix pour mesurer quantitativement des quantités de départ de l'ADN, un ADNc ou un ARN. Q-PCR est généralement utilisé pour déterminer si une séquence d'ADN est présente dans un échantillon et le nombre de ses copies dans l'échantillon. La méthode avec actuellement le plus haut niveau de précision est la PCR quantitative en temps réel. Il est souvent connu sous le nom de prêter à confusion RT-PCR (R eal T ime PCR) ou RQ-PCR. QRT-PCR ou RTQ-PCR sont des contractions plus appropriés. RT-PCR se réfère communément à transcription inverse PCR (voir ci-dessous), qui est souvent utilisé en conjonction avec Q-PCR. Méthodes QRT-PCR utilisent des colorants fluorescents, tels que les SYBR Green, ou des sondes d'ADN contenant un fluorophore, tels que TaqMan, pour mesurer la quantité de produit amplifié en temps réel.
- RT-PCR: (Réverse T ranscription PCR) est une méthode utilisée pour amplifier, isoler ou identifier une séquence connue d'un cellulaire ou tissu ARN. La PCR est précédée d'une réaction en utilisant transcriptase inverse pour convertir ARN à ADNc. RT-PCR est largement utilisé dans le profilage d'expression, afin de déterminer l'expression d'un gène ou d'identifier la séquence d'un transcrit d'ARN, y compris le démarrage de la transcription et des sites de terminaison et, si la séquence d'ADN génomique d'un gène est connue, pour cartographier l'emplacement du exons et introns dans le gène. L'extrémité 5 'd'un gène (par rapport au site d'initiation de la transcription) est généralement identifié par un procédé de RT-PCR, nommée RACE-PCR, court pour l'amplification rapide des extrémités d'ADNc.
- Solid Phase PCR: englobe de multiples significations, y compris Polony Amplification (où les colonies PCR sont dérivés dans une matrice de gel, par exemple), 'Bridge PCR »(les seules amorces présente sont liés de manière covalente à la surface du support solide), en phase solide classique PCR (où PCR asymétrique est appliquée en présence de solide soutien amorce de roulement avec une séquence correspondant à l'une des amorces aqueuses) et Enhanced solide PCR phase (où phase solide conventionnelle PCR peut être améliorée en utilisant haute Tm amorce de support solide avec application d'une 'étape' thermique à favoriser support solide amorçage).
- TAIL-PCR: PCR asymétrique thermique entrelacée est utilisée pour isoler une séquence inconnue flanquant une séquence connue. Dans la séquence connue TAIL-PCR utilise une paire d'amorces imbriquée avec différentes températures de recuit; une amorce dégénérée est utilisée pour amplifier dans l'autre direction à partir de la séquence inconnue.
- Touchdown PCR: une variante de la PCR qui vise à réduire de fond non spécifique en abaissant progressivement la température de recuit que le cyclisme PCR progresse. La température de recuit à des premiers cycles est généralement de quelques degrés (3-5c) au-dessus de la Tm des amorces utilisées, tout en les cycles ultérieurs, il est de quelques degrés (3-5c) ci-dessous l'amorce T m. Les températures plus élevées donnent une plus grande spécificité pour la liaison à l'amorce, et les températures inférieures permettent une amplification plus efficace des produits spécifiques formés pendant les cycles initiaux.
- PAN-AC: Cette méthode utilise pour l'amplification des conditions isothermes, et peut être utilisé dans les cellules vivantes.
- Universal marche rapide: cette méthode permet marche du génome et l'empreinte génétique en utilisant un plus spécifique «recto-verso» PCR que les approches conventionnelles «unilatérales» (en utilisant seulement une amorce spécifique du gène et une amorce générale - qui peut conduire à «bruit artefactuelle ') en vertu d'un mécanisme impliquant la formation de structure de type lariat. Rationalisation des dérivés de UFW sont Lane RAGE (lasso termine dépendant PCR nichée pour l'amplification rapide de l'ADN génomique), 5'RACE Lane et 3'RACE Lane.
Histoire
Un document 1971 dans la Journal of Molecular Biology par Kleppe et collaborateurs décrit un premier procédé utilisant un dosage enzymatique pour répliquer une matrice d'ADN avec des amorces courte in vitro. Toutefois, cette manifestation précoce du principe de base de PCR n'a pas reçu beaucoup d'attention, et l'invention de la réaction en chaîne de la polymérase en 1983 est généralement crédité à Kary Mullis.
Au cœur de la méthode PCR est l'utilisation d'un convenable ADN polymérase capable de résister aux hautes températures de> 90 ° C (> 195 ° F) requise pour la séparation des deux brins d'ADN dans le Double hélice de l'ADN après chaque cycle de réplication. Les ADN polymerases utilisées initialement pour présageant expériences in vitro par PCR ont été incapables de résister à ces températures élevées. Ainsi, les premières procédures de replication de l'ADN ont été très inefficaces, de temps, et doivent de grandes quantités d'ADN polymerase et de manutention continue tout au long du processus.
Une découverte de 1976 Une polymerase Taq ADN polymérase purifiée à partir de la bactérie thermophile, Thermus aquaticus, qui se produit naturellement dans l'eau chaude (50-80 ° C (120-175 ° F)) environnements ouvert la voie à des améliorations spectaculaires de la méthode PCR. La polymerase d'ADN isolé à partir de T. aquaticus est stable à des températures élevées en restant actif même après dénaturation de l'ADN, ce qui évite la nécessité d'ajouter de nouveaux ADN polymérase après chaque cycle. Cela a permis un processus fondé de thermocycleur-automatisé pour l'amplification d'ADN.
Au moment où il a développé PCR en 1983, travaillait dans Mullis Emeryville, en Californie, pour Cetus Corporation, l'un des premiers biotechnologie entreprises. Là, il était responsable de la synthèse de courtes chaînes d'ADN. Mullis a écrit qu'il a conçu de la PCR en croisière le long de la Pacific Coast Highway une nuit dans sa voiture. Il jouait dans son esprit avec une nouvelle façon d'analyser les changements (mutations) dans l'ADN quand il a réalisé qu'il avait plutôt inventé un procédé d'amplification d'une région de l'ADN par des cycles répétés de duplication entraînée par l'ADN polymérase.
En Scientific American, Mullis résumé la procédure: "A partir d'une seule molécule de l'ADN du matériel génétique, le PCR peut générer 100 milliards de molécules semblables dans un après-midi La réaction est facile à exécuter Il ne nécessite pas plus d'un tube à essai, quelques-uns.. réactifs simples, et une source de chaleur. " Il a reçu le prix Nobel de chimie en 1993 pour son invention, sept ans après que lui et ses collègues de Cetus première mise sa proposition à la pratique. Cependant, certaines controverses sont restés sur les contributions intellectuelles et pratiques d'autres scientifiques aux travaux de Mullis, et se il avait été le seul inventeur du principe PCR. (Voir l'article principal: Kary Mullis)
Guerre des brevets
La technique PCR a été breveté par Cetus Corporation, où Mullis a travaillé quand il a inventé la technique en 1983. L'enzyme Taq polymérase a également été couverts par des brevets. Il ya eu plusieurs procès très médiatisés liés à la technique, y compris une poursuite intentée par infructueuse DuPont. La compagnie pharmaceutique Hoffmann-La Roche a acheté les droits sur les brevets en 1992 et détient actuellement ceux qui sont encore protégés.
Une bataille de brevet liées sur l'enzyme Taq polymérase est toujours en cours dans plusieurs pays à travers le monde entre Roche et Promega. Les arguments juridiques ont étendu au-delà de la durée de vie des brevets de la polymérase Taq PCR et originaux, qui a expiré le 28 mars, 2005
